天昊生物微生物扩增子测序数据分析全面升级!
QIIME(Quantitative Insights Into Microbial Ecology)是于2010年发表于Nature Methods的一款进行微生物群落结构分析的开源生物软件,是微生物组研究领域使用最广泛的扩增子测序数据分析流程。
而于2018年全面接替QIIME的QIIME 2,是一个经过完全重新设计和重写的系统(https://qiime2.org/)(源代码见 https://github.com/qiime2),可实现对微生物组数据的可重复性和模块化分析,是代表未来趋势的微生物组分析方法最新标准,相关结果于2019年7月已经发表于顶级期刊Nature Biotechnology[1]!
QIIME2优点
1)生成的图表文件类型.qzv 保证了分析结果的交互式可视化2)统一的分析过程文件格式.qza保证了每一步分析结果可重复,可追溯3)支持自定义分析插件加入分析流程保证了系统功能的可扩展性4)安装非常方便5)分析流程化标准化6)使用方式更加多样
图1 QIIME2提供了许多交互式可视化工具
a,未加权UniFrac主坐标分析图,包含37680个样本,说明了QIIME 2的大样本量处理能力。颜色表示样本类型,如地球微生物组计划本体(EMPO)所述。
b,交互式物种分类组成条形图,显示了沿黄石国家公园温泉流出通道中的不同温度梯度收集的微生物样品的门水平物种组成,该图中可用的许多交互式控件大大降低了QIIME 1的探索性分析负担。
c,特征挥发性图,说明了母乳喂养和配方喂养的婴儿中双歧杆菌丰度随时间的变化。通过此可视化可以交互式地发现时空特异的特征菌,条形图对重要性(时间点的预测能力)和微生物丰度进行排序。这些条形图提供了一个界面,用于根据其重要性和丰度可视化单个特征菌的折线图,点击条形图将显示该特征菌的折线图,并在下面的条形图中以蓝色突出显示该特征菌的重要性和丰度。
d,人类皮肤表面的分子地图。 彩色斑点表示小分子化妆品成分月桂基硫酸钠在人体皮肤上的含量。 样本数据可以在三维模型中进行交互可视化,从而支持空间模式的发现。
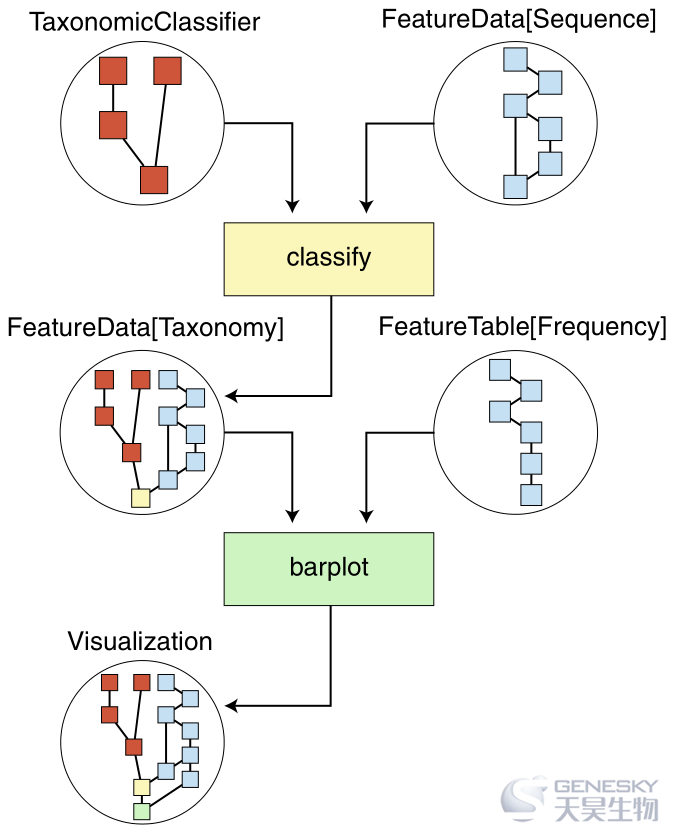
图2 QIIME2迭代记录数据来源,确保生物信息学分析的再现性
此简化图说明了有关图1c中显示的物种分类组成条形图创建的自动跟踪信息。 QIIME 2结果(圆圈)包含网络图,该网络图说明了存储在结果中的数据来源。动作(四边形)应用于QIIME 2结果并产生新结果。箭头指示通过行动进行的QIIME 2结果的流程。TaxonomicClassifier和FeatureData [Sequence]输入包含独立的出处(分别为红色和蓝色),并提供给分类动作(黄色),在分类学上注释序列。分类动作的结果FeatureData [Taxonomy]将两个输入的来源与分类动作整合在一起。然后通过FeatureTable [Frequency]输入将该结果提供给barplot动作,该输入与FeatureData [Sequence]输入共享一些出处,因为它们是从同一上游分析生成的。产生的可视化效果(图1c)具有完整的数据来源,并正确标识了输入的共享处理。
图S1 QIIME 2系统的示意图
接口定义了用户与系统的交互方式;插件定义了所有特定域的功能;框架介导插件和接口之间的通信,并执行核心功能,如出处跟踪。箭头指示依赖性。
接口仅与qiime2.sdk子模块交互,而插件仅与qiime2.plugin子模块交互。 这种设计可以方便第三方插件和界面开发人员扩展这个系统。
图S2 QIIME 2与接口无关
全面的QIIME 2功能对于计算机水平不同的研究人员来说都是可用的,这是与提供单一接口的QIIME 1等技术相比的一大优势。
(a)想要查看QIIME 2结果或数据来源的用户可以在不安装QIIME 2的情况下使用QIIME 2 View,这对于希望研究其他人产生的交互式可视化结果的首席研究人员,临床医生或政策制定者很方便。
(b)喜欢图形界面的研究人员可以使用原型图形界面QIIME 2 Studio。 这对于没有命令行或编程技能的用户来说很方便。
(c)熟悉Linux命令行和/或经常在机构计算机集群上工作的用户可以通过命令行界面q2cli使用QIIME 2。(d)数据科学家(例如,在Jupyter Notebooks中工作的用户或对自动化QIIME 2工作流程感兴趣的用户)可以通过Python 3“artifact API”使用QIIME 2。
QIIME2流程之DADA2插件
因为目前QIIME2流程有很多插件,下面就为大家着重介绍其中的明星插件DADA2。QIIME2就是采用DADA2插件对原始数据进行降噪,拼接及去嵌合体,其质控条件更严格,仅去重复序列,不再对序列进行相似度聚类,用扩增序列变体(amplicon sequence variant,ASV)替代操作分类单元(Operational Taxonomic Units,OTU),提高了分类学分辨率,得到结果更加真实可靠,为什么这么说呢,传统的OTUs相似聚类阈值是97%,而DADA2分析则是以100%的相似度聚类,2016年发表在Nature Method [2]上的相关研究结果表明,DADA2分析可以获得单碱基精度的代表序列,这不但大大提高了样本中真实的种甚至菌株水平的鉴别率,同时降低假阳性OTUs的几率,这对后续开展特定单菌的功能验证实验打下坚实可靠的基础。
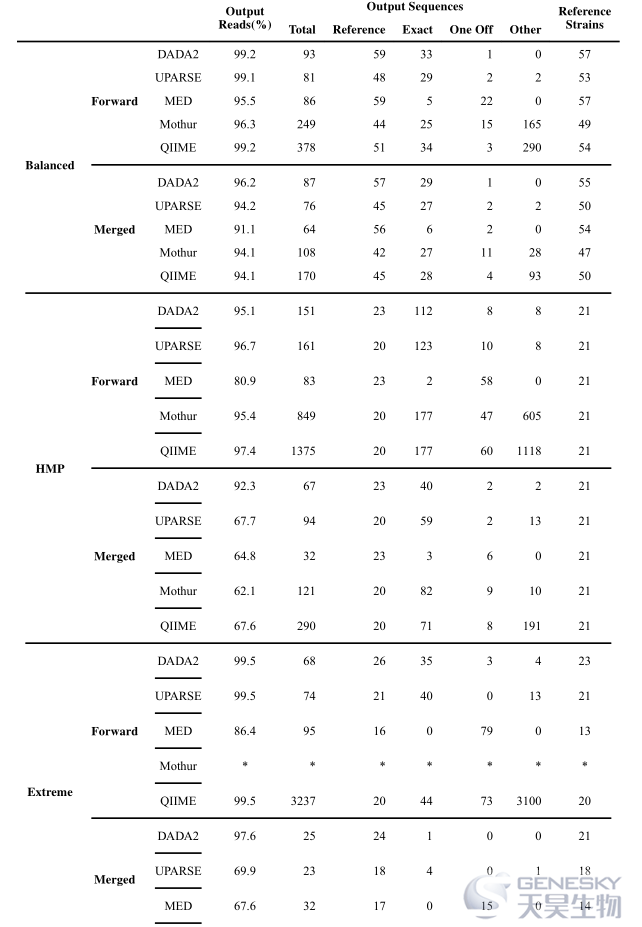
表1 通过与这些模拟群体的已知序列(参考菌株)进行比较,并与核酸序列进行比较以鉴定污染物,将输出序列分为Reference 或者 Exact(真阳性)和One Off 或者Other(假阳性)。 结果发现DADA2检测到最多的参考菌株和序列,同时输出最少的假阳性。
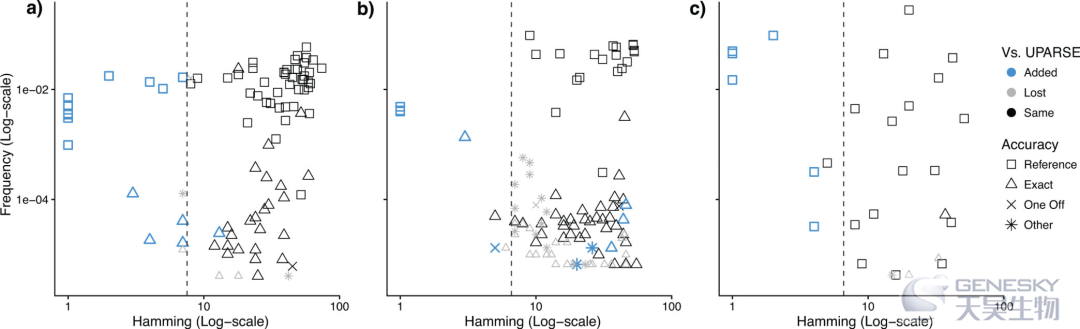
图1 DADA2推断的序列变体与UPARSE构建的OTUs比较。结果表明DADA2推断的序列变体与UPARSE输出的OTUs(黑色)基本一致,但是,DADA2发现了额外真实的物种(蓝色),同时输出了更少的假阳性序列(One Off and Other)。
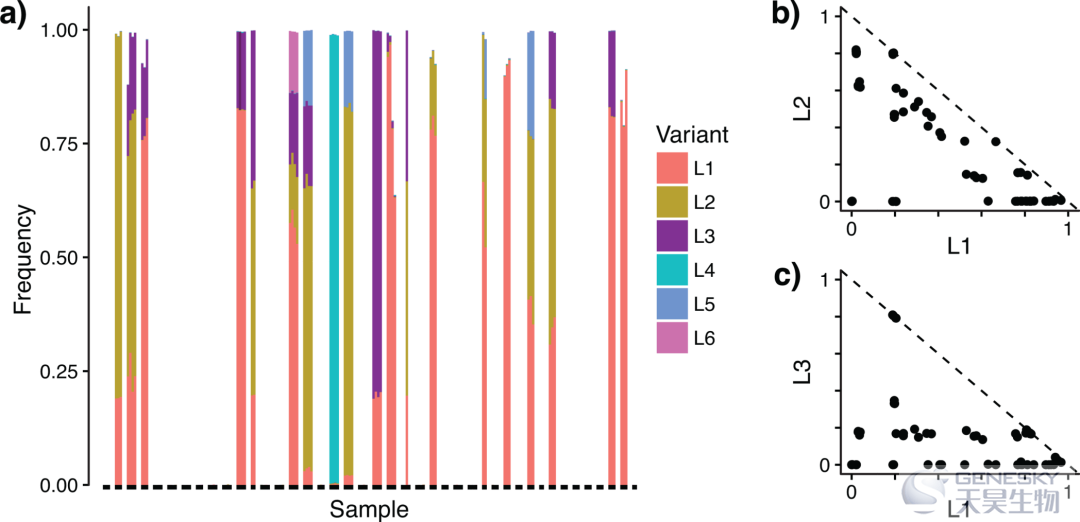
图2 怀孕期间人类生殖道群落中的Lactobacillus crispatus序列变体。结果表明DADA2可以鉴定到多个样品中原本存在但是传统OTUs聚类没有发现的六个乳杆菌16S rRNA序列变体。
参考文献:
1、Bolyen E, Rideout JR, Dillon MR, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nature Biotechnology. 2019 Aug;37(8):852-857.
2、Callahan BJ, McMurdie PJ, Rosen MJ, et al. DADA2:high-resolution sample inference from Illumina amplicon data. Nature methods, 2016 Jul;13(7):581-3.
看完上述描述,是不是感觉QIIME2平台很强大呢,是不是特别想尝试一下这一革命性的创新分析流程呢,不用着急,天昊生物紧跟生信分析新趋势,已将公司原有的相对定量扩增子数据分析流程和绝对定量扩增子数据分析流程全面升级为基于QIIME 2的分析流程,从而让老师们的研究成果符合行业最新标准,能够更快更好的发表研究成果,心动不如行动,欢迎各位老师与我们沟通联系!

 咨询热线:400-065-6886
咨询热线:400-065-6886

 咨询热线:400-065-6886
咨询热线:400-065-6886